

Des recommandations nationales permettent une systématisation de la classification des anomalies identifiées lors du séquençage de l’ADN de patients atteints de maladies génétiques.
Les maladies génétiques sont dues aux anomalies de l’ADN, des molécules qui sont présentes dans toutes les cellules de notre corps et qui portent l’information génétique. L'identification de ces anomalies est un véritable défi, car l’ensemble de notre patrimoine génétique, ou génome, est composé de trois milliards de nucléotides (les « lettres » de notre code génétique), dont l’agencement forme notamment les 20 000 gènes comportant l’information pour la synthèse des protéines qui constituent nos cellules.
Pour détecter des anomalies de notre génome, la mise en place diagnostique de nouvelles technologies de séquençage (aussi appelées NGS, « Next Generation Sequencing ») a amené une véritable révolution dans le domaine de la Génétique médicale, car elles permettent de séquencer, ou « lire », de grandes parties de nos génomes et d’identifier les différences de la séquence génomique (ou « variants ») chez un patient, par rapport aux données de génomes de référence d’individus sains disponibles dans des bases de données internationales.
Puisqu’il existe de manière naturelle et non pathologique une grande variabilité génétique entre individus (la séquence du génome de deux individus non apparentés diffère en moyenne de 0,1%, soit 3 millions de nucléotides différents entre vous et votre collègue), le défi majeur qui se présente est de distinguer les variants « pathogènes », de simples variants de la normale impliqués dans la diversité physiologique entre individus.
Cet enjeu de distinguer le pathologique du normal est comparable à celui rencontré en imagerie médicale, avec une conséquence majeure sur le diagnostic de précision et ainsi la prise en charge du patient.
Pour retenir le caractère pathogène d’un variant de l’ADN, il faut ainsi recueillir de nombreuses informations (type de variant, transmission familiale, récurrence du variant chez des individus atteints, existence du variant dans la population générale ou non…).
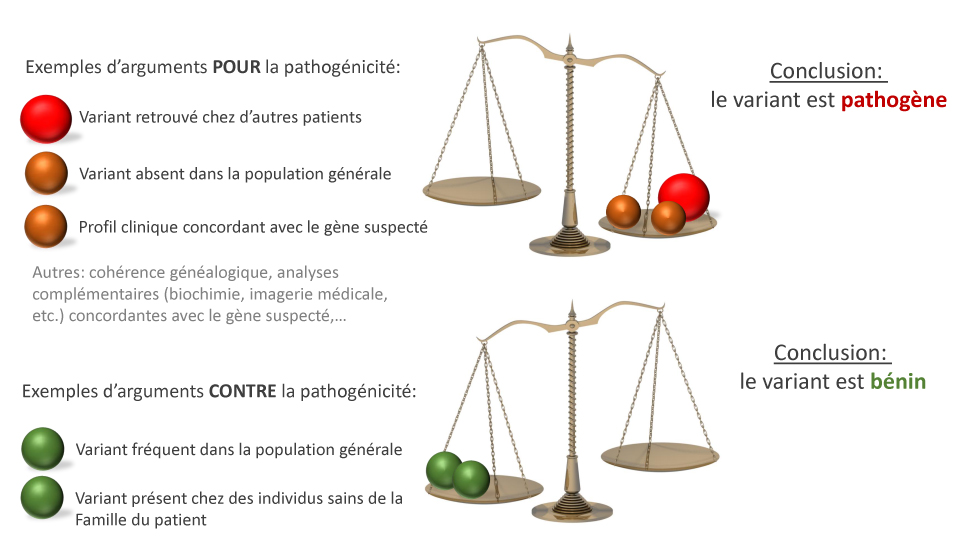
Afin d’homogénéiser et standardiser l’interprétation clinique des variants entre les laboratoires diagnostiques, des recommandations internationales ont été proposées en 2015 par l’ACMG/AMP (American College of Medical Genetics & Association for Molecular Pathology).
Sous l’égide d’un groupe de travail du Réseau NGS-Diag coordonné par le Dr. Pascale SAUGIER-VEBER (CHU Rouen), le Dr. Svetlana GOROKHOVA-DEVRED et le Pr. Martin KRAHN (AP-HM, CHU Marseille, Service du Pr. Nicolas LÉVY), les recommandations de l’ACMG ont été adoptées en 2018 et actualisées récemment (diffusion par la Fédération Française de Génétique Humaine [3]) sous forme de recommandations nationales validées par toutes les sociétés savantes françaises impliquées dans le diagnostic génétique, dans le domaine des maladies rares et des cancers constitutionnels. Ces recommandations sont utilisées dorénavant dans les laboratoires hospitaliers de génétique en France, ainsi que sur les plateformes nationales du Plan France Médecine Génomique [4].
A Marseille, les membres du Service de Génétique médicale participent depuis de nombreuses années à différentes activités de coordination nationales, dont l’objectif est notamment une extension de l’accès au séquençage à haut débit dans le parcours de soin des patients. Dans ce cadre, notre institution bénéficiera dans les prochaines années de la mise en place de l’Institut euroméditerranéen de lutte contre les maladies rares génétiques GIPTIS [5], qui constituera un environnement d’exception pour le développement de projets innovants dans ce domaine.
Links
[1] https://fr.ap-hm.fr/print/819792
[2] https://fr.ap-hm.fr/printpdf/819792
[3] http://ffgh.net/index.php/presentation/les-reseaux-partenaires/reseau-ngs-diagnostic/182-reseaux/ngs/354-recommandations-professionnelles-du-reseau-ngs-diag
[4] https://solidarites-sante.gouv.fr/systeme-de-sante-et-medico-social/recherche-et-innovation/france-genomique
[5] http://www.giptis.com
[6] https://www.facebook.com/sharer/sharer.php?u=https://fr.ap-hm.fr/print/819792
[7] https://www.linkedin.com/shareArticle?mini=true&url=https://fr.ap-hm.fr/print/819792&title=Maladies génétiques : homogénéisation nationale de l’interprétation des résultats issus du séquençage à haut débit
[8] https://twitter.com/intent/tweet/?text=Maladies génétiques : homogénéisation nationale de l’interprétation des résultats issus du séquençage à haut débit&url=https://fr.ap-hm.fr/print/819792
[9] mailto:?subject=Maladies génétiques : homogénéisation nationale de l’interprétation des résultats issus du séquençage à haut débit&body=https://fr.ap-hm.fr/print/819792